Después de una semana intensa, ganamos la “Emprendethon”, evento donde nos pasaron juguetitos nuevos con los que armamos dos prototipos de aplicaciones.
En este artículo, describiremos de qué trató este evento, cuáles fueron esas dos aplicaciones que construimos, qué ganamos y nuestras impresiones generales de la jornada.
¿Qué es la Emprendethon?
Durante la semana del 9 al 13 de enero, se realizaron en la ciudad de Valparaíso, Chile, dos importantes eventos para el mundo de la salud digital, la Connectathon y el Make Health Latam.
La Connectathon, estuvo enfocada a que distintos grupos pudiesen aplicar el uso del estándar HL7 FHIR, utilizando los perfiles adaptados para nuestro país.
En el Make Health, hubo charlas, paneles, workshops y una Datathon, en dónde los equipos estuvieron enfocados en utilizar bases de datos entregadas por diferentes organizaciones, para hacer Ciencia de Datos.
La Emprendethon funcionó de forma transversal a estos 2 eventos, y consistía en que emprendedores aprendieran a utilizar distintas herramientas en el ámbito de interoperabilidad y ciencia de datos, pero no solo desde el punto de vista técnico/académico, sino que también mostrar cómo lo vería un usuario ya sea profesional de salud o paciente, intentando transmitir la cadena de valor completa que nos entregan esos datos.
Connectathon
El desafío durante la Connectathon (días 1 y 2)
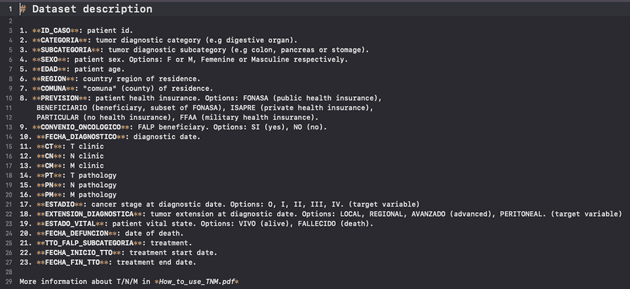
Para el primer desafío, bajo el alero de la Connectathon, se nos entregó un caso de uso donde el objetivo era que lográsemos guardar y consultar información de un paciente y sus diagnósticos.
Obviamente, los datos tenían que cumplir con estándares de interoperabilidad, para lo cual nos dieron acceso a un servidor FHIR disponibilizado por AWS, el cual contaba con las reglas de la guía core de Chile.
Además, utilizando algunas herramientas de AWS, debíamos construir una aplicación web que se conectara al servidor FHIR y consultara los datos almacenados. La herramienta debía ser publicada en una URL de manera de que cualquier persona pudiera visualizarla.
Nuestra solución
Lo primero que hicimos fue extraer la información del caso de uso e identificar los recursos FHIR que podían servirnos para almacenar esta información. En este repositorio GitHub están los archivos JSON, con los recursos que logramos armar. Por temas de tiempo y de las definiciones que nos entregaron para esta situación, hubo varios que finalmente no utilizamos.
Una vez guardados los datos en el servidor, siguiendo las definiciones del estándar, llegó el momento de construir la aplicación.
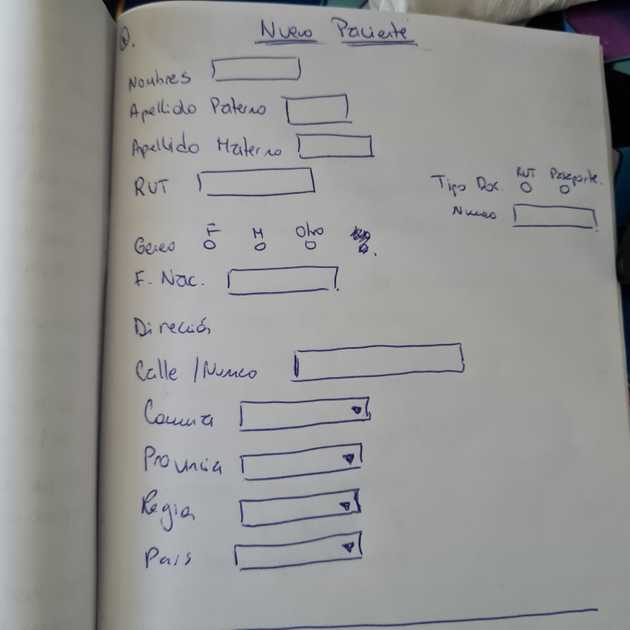
Nuestro diseño y planificación inicial tenía 3 partes:
- Un buscador, que recibiera el rut del paciente, y que se encargará de ir a “preguntar” al servidor por la información.
- Una vista de resumen de paciente, que mostrara los datos traídos de la consulta anterior.
- Otra pantalla que tuviese un formulario, para poder crear un paciente, o al menos agregarle un nuevo diagnóstico al paciente existente.
Como suele suceder cuando se crean soluciones tecnológicas, el tiempo no era el suficiente y nos tocó priorizar. Es decir, para armar nuestro MVP (producto mínimo viable, por sus siglas en inglés) tuvimos que preguntarnos cuál de todos estos puntos entregaba más valor y era el más esencial. La respuesta fue: la vista de resumen con los datos del paciente ya guardados. Así que ese fue el objetivo inicial, si nos quedaba más tiempo íbamos a ir por el buscador y luego el formulario.
Por supuesto no nos quedó más tiempo, al finalizar el segundo día, logramos desplegar la aplicación con la vista de resumen del paciente solamente.
Detalles técnicos
Si no entiendes mucho de términos específicos de desarrollo web, puedes saltarte esta sección que podría sonar a chino y pasar a la siguiente sin perderte nada, de todos modos intentaré explicarlos lo más en “humano” posible.
El recurso Patient, que como se puede deducir hace alusión al paciente, lo armamos siguiendo las definiciones y ejemplos de la guía para Chile.
Un tip interesante a recordar de la guía core de Chile es que el atributo Address se reemplaza por CL Address, que es igual, pero incluye extensiones obligatorias (city, district, state y country que para nosotros sería: comuna, provincia, región y país respectivamente).
La idea del ejercicio era que también utilizáramos el recurso Bundle, que en resumen permite agrupar y crear al mismo tiempo varios recursos relacionados en este caso con el mismo paciente, es por eso que en el detalle se puede ver que contiene varios recursos Condition, que son los diagnósticos del paciente.
Una vez creado este Bundle, que como ya comentamos en el post anterior, nos demandó más tiempo del planificado, porque no lográbamos que el servidor aceptara la información. Llego el momento de construir la aplicación.
Para desarrollar, por temas de velocidad y conocimiento de ambos, decidimos utilizar el framework de desarrollo fron-end React y le pusimos un poquito de estilo con la librería de interfaz de usuario Ant Design.
Para realizar el despliegue utilizamos 2 herramientas de AWS:
- AWS Amplify, para hostear la aplicación.
- AWS CodeCommit, que nos permitía desplegar cambios con solo hacer push a la rama main.
Cuando desplegamos la aplicación realizando la consulta por los datos del paciente al servidor FHIR, nos encontramos con un problema importante de seguridad, la aplicación desplegada en Amplify no admitía realizar una consulta a un servidor que no fuera https, y el que nos entregaron para trabajar solo funcionaba con http 😞.
Googleando, logramos encontrar librerías que hacían de proxy para forzar este tipo de llamadas, pero ninguna nos funcionó. Luego Nico dio con el código open source de una (cors-anywhere) y levantó su propio proxy que si dio resultado 🎉 (todo esto último lo hicimos en unos 20 minutos 😅).
Datathon
El desafío durante la Datathon (días 3 y 4)
El tercer día comenzó el evento Make Health y con este la Datathon. El día inició un poco confuso para nosotros, ya que por llegar “heredados” de la Connectathon ni siquiera tenían registro de nuestros nombres en la entrada 😂.
Una vez ingresamos, similar al primer día, tuvimos un par de charlas que se extendieron hasta bastante avanzada la tarde, así que comenzamos a revisar la información para trabajar cerca del final del día.
En este caso se nos disponibilizaron datos del Registro Hospitalario de Tumores de la FALP (Fundación Arturo López Pérez), la idea era que utilizando herramientas de AWS lográsemos trabajar con esos datos, analizarlos y quizás lograr crear un modelo predictivo.
Nuevamente, tal como se planteó en el desafío anterior, se debía crear una aplicación que pudiese interactuar de alguna manera con todos estos datos trabajados.
La solución
La primera alternativa era limpiar los datos, analizarlos y buscar una forma de que estos datos “ordenaditos” se mostraran de una forma amigable para un usuario, por ejemplo, del ámbito clínico. No nos gustó mucho la idea, porque queríamos jugar más con las herramientas de AWS (no todos los días se puede acceder gratis a estas cosas).
Otra opción, y con esta alternativa podíamos utilizar más los conocimientos de Nico en Data Science, fue intentar hacer un modelo predictivo que se entrenara con los datos que nos entregaron. Para esto me puse estudiosa, a tratar de refrescar los conocimientos adquiridos en fisiopatología y epidemiología, para intentar buscar una predicción que pudiese resultar de valor. Una idea loca que surgió fue intentar predecir la sobrevida de un paciente con cáncer según la etapa en la que se diagnóstica y el tipo de tratamiento que recibe, era un tema interesante, pero requería más tiempo.
Así que finalmente nos decidimos por algo un poco más simple, y que más que nada sirviera de prueba de concepto de las herramientas que estábamos probando. Por lo que decidimos crear un formulario que recibiera cierta información demográfica del paciente e información del cáncer diagnosticado, y que predijera el estadio de este cáncer. (Para recordar todo esto de las etapas y el estadio me sirvió mucho, entre otra documentación, esta página: cancer.org).
Detalles técnicos
En este desafío para poder llegar a tiempo nos tocó dividirnos según experiencia y conocimiento, así que, por un lado, Nico se dedicó a construir el modelo predictivo y por mi parte, armé la aplicación con el formulario de ingreso de datos.
Con respecto al modelo predictivo, no partimos de cero. Los datos entregados por la FALP no eran solo bases de datos, sino que también incluían instrucciones de limpieza y de exploración. Por ejemplo, había archivos (Jupyter Notebooks) donde analizaban los datos:
También incluía una descripción del Dataset:
Y código para limpiar:
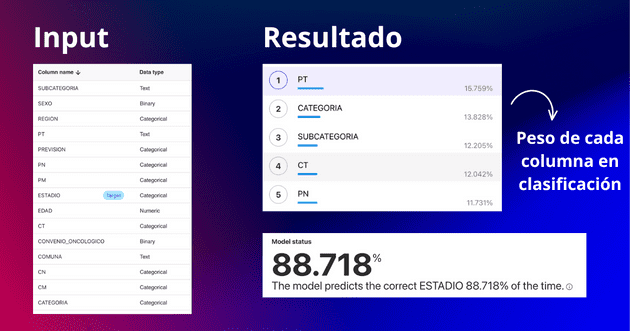
A base de todo lo anterior, pudimos notar que el problema es de clasificación, en donde dado ciertos atributos demográficos del paciente como género, edad y comuna, hay que predecir en qué estadio de cáncer se encuentra.
Para continuar con la creación del modelo, seguimos los siguientes pasos:
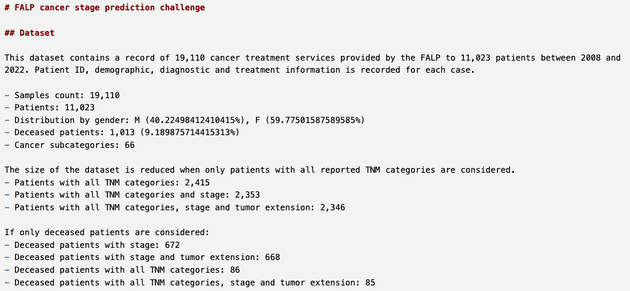
- Análisis de los datos: el conjunto de datos era un CSV con 19.000 filas y 23 columnas. Los archivos que nos entregaron incluían una pequeña exploración, donde indicaba la cantidad de pacientes, distribución según género, entre otros aspectos. Nos quedamos con este análisis por temas de tiempo, aunque nos hubiese encantado hacer uno más profundo.
- Limpieza de los datos: el dataset venía con instrucciones para limpieza e incluso código en Python, así que no fue tan complicado limpiarlos. Nos quedamos con 2.346 filas, y varias columnas menos. Por ejemplo, no usamos información del tratamiento, ya que esto es algo que ocurre después de la clasificación de estadio.
- El modelo predictivo: aquí usamos dos herramientas de Amazon. Una fue SageMaker Canvas y la otra SageMaker Autopilot. SageMaker Canvas fue un primer acercamiento, en menos de 15 minutos nos creó un modelo con 89% de precisión y nos decía qué atributos eran más importantes. Lo malo es que no encontramos una opción para poder usarlo fuera de la herramienta. Así que nos cambiamos a SageMaker Autopilot, que demoraba más en entrenar (~40 minutos) pero nos dejaba un modelo disponible para ser usado.
- La puesta en marcha del modelo: Elegimos el mejor modelo sugerido por SageMaker según la precisión (no encontramos cómo cambiar la métrica), y con 1 clic lo pusimos en ejecución.
- Disponibilizando el modelo para la web: Gracias a la sugerencia de los “amigos de Amazon” (mentores), logramos hacer que el modelo estuviera disponible a través de internet. Esto lo hicimos con una función Lambda (código fuente), que respondía a eventos HTTP y que se comunicaba con el modelo creado con SageMaker.
De esta imagen, podemos ver que el input consiste en 15 columnas, y que el resultado fue 88.7 precisión, con PT, categoría y sub-categoría como campos más importantes. Posiblemente hay cosas por mejorar, como por ejemplo, el par comuna y región quizás no es necesario, ya que la región se infiere de la comuna. Es posible que pase lo mismo con el par categoría y sub-categoría.
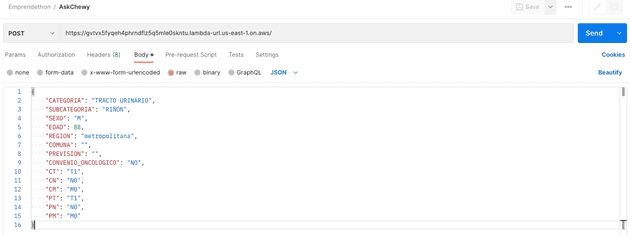
Gracias a lo anterior, ya podíamos hacer solicitudes HTTP para hacer predicciones. Por ejemplo, con Postman:
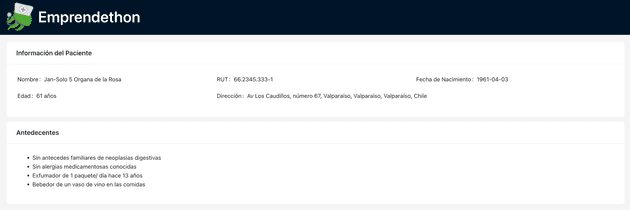
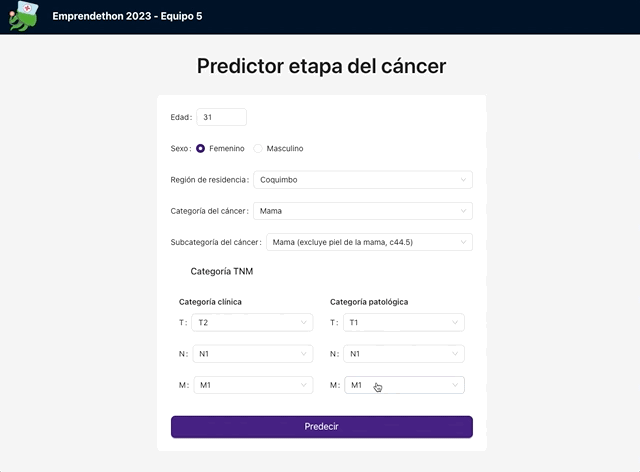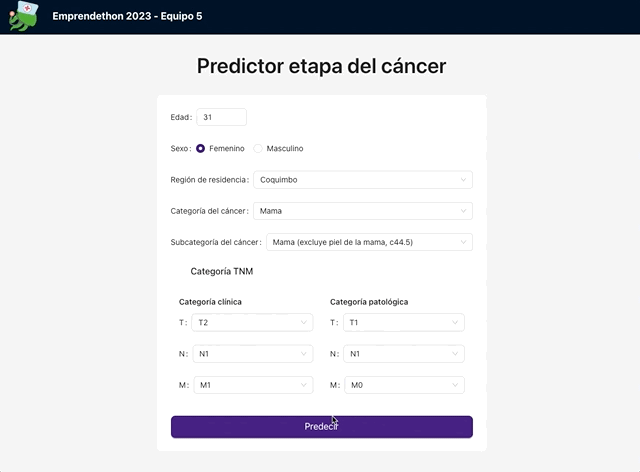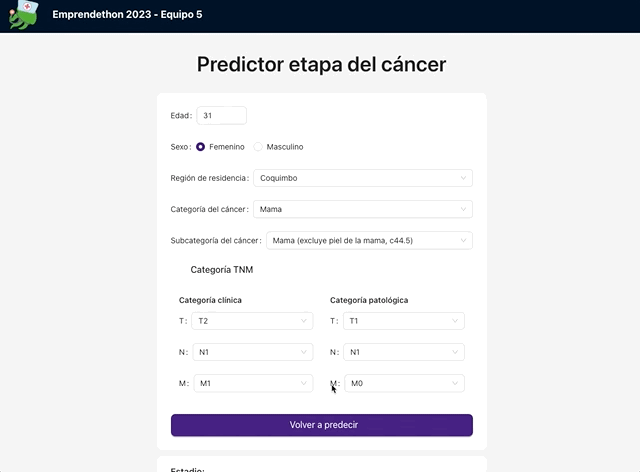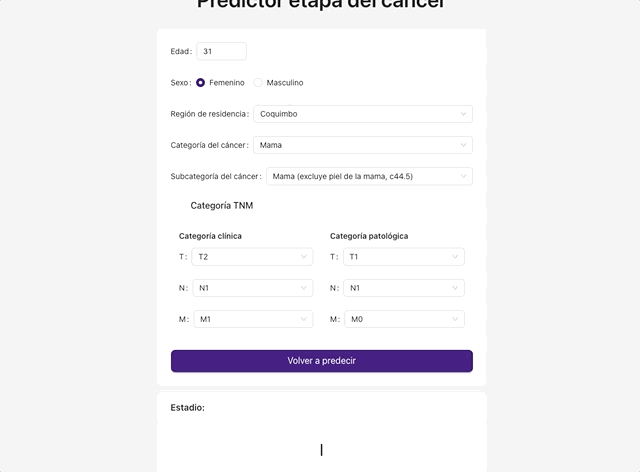
En cuanto a la aplicación, tal como la anterior, la construimos en React y utilizamos para la interfaz de usuario Ant Design. Los campos disponibles en el formulario eran:
- Edad
- Sexo
- Región de residencia
- Categoría del cáncer
- Subcategoría del cáncer
- Categoría TNM clínica (Tumor-Nódulo-Metástasis)
- Categoría TNM patólogica (Tumor-Nódulo-Metástasis)
Le puse cariño en la construcción del formulario para que además de verse decente mostrara las diferentes alternativas de forma bonita para el usuario, aunque “por detrás” mapeaba cada respuesta a cómo el modelo esperaba recibirlas. Esto lo hice creando “diccionarios” en formato JSON, que se encargaban de transformar cada respuesta.
Ejemplo del cómo se transformaban las regiones:
La parte que requirió más detalle fue lograr clasificar las sub-categorías, de tal manera de que una vez seleccionada la categoría principal, solo le permitiera al usuario seleccionar las sub-categorías correspondientes.
Ejemplo del cómo se agrupaban las sub-categorías dentro de cada categoría:
Para terminar, conectamos esta aplicación con el endpoint que Nico había generado. Es decir, envíabamos los datos recogidos por el formulario a ese endpoint, para que estos fueran procesados por el modelo y este nos entregara como respuesta el estadio del cáncer. Finalmente, la aplicación le mostraba al usuario este resultado.
El siguiente GIF muestra el resultado final:
¿Qué ganamos?
El honor y la gloria de ganar un evento como estos ⚔️, junto a un iPad y un curso para cada uno para seguir aprendiendo sobre HL7 🤓.
Impresiones generales
Como conclusiones finales de estos agotadores, pero gratificantes 4 días de trabajo, podemos decir que fue emocionante ver a tanta gente reunida en la misma sintonía, buscando cada uno desde su lugar impactar en mejorar la salud con el apoyo de la tecnología. Como enfermera, cuando estaba en el mundo clínico me tocaba ver estos conceptos como algo totalmente lejano, y es esperanzador ver que hoy ya son una realidad.
Por eso este tipo de instancias, con espacio para ir a escuchar, pero también para participar activamente y “ensuciarse” las manos, permite no solo aprender, sino que también compartir el conocimiento y las experiencias, de sea cual sea el rol que tienes en la salud (profesional clínico, informático, paciente, familiar).
Un aspecto súper positivo fue el apoyo que se consiguió de AWS, ya que no solo nos facilitaron cuentas para utilizar gratis sus diferentes herramientas durante el periodo del evento, también estaban los “amigos Amazon” como los llamamos con Nico, monitores que nos enseñaron en simples pasos a usar las herramientas y nos resolvían cualquier duda que tuviésemos sobre ellas.
En cuanto a las herramientas de AWS, nos llevamos una grata sorpresa. Amplify se ve muy robusto para crear aplicaciones, mientras que las herramientas de SageMaker nos facilitaron mucho la vida a la hora de crear el modelo predictivo, nos resultó infinitamente más sencillo que haber creado nosotros mismos los modelos.
Nos hubiese encantado participar de todas las instancias, las charlas, los paneles, el espacio de la innovación, pero estábamos sumergidos en intentar hacer lo mejor posible en el track que elegimos. Así que ahora nos queda la tarea de ver las grabaciones.
Como en cualquier situación de la vida, hubo cosas mejorables, pero lo más importante fue que los organizadores lograron hacer esto realidad. Había momentos en que estuvimos súper perdidos sin saber cómo continuar, y qué debíamos entregar, pero con Nico nos reíamos pensando en que por algo se llamaba “Emprendethon”, cuando emprendes todo es medio incierto, no necesariamente sabes lo que tienes que hacer, y así estábamos nosotros.
Sentimos que logramos el objetivo, partimos intentando plasmar cómo se recogen los datos ingresados por un usuario, se guardan estandarizados y luego se consultan para su uso primario. Después, continuamos con lo que se puede hacer si esos datos se procesan y se utilizan para crear herramientas que aporten en cierta forma a mejorar algún aspecto de la salud.